- 首页
- 关于我们
-
产品中心
- 光片显微镜系列
- LS-COMPACT光片显微镜 LS18平铺光片显微镜 锘海组织透明化底透台 透明度检测仪 组织透明化染色仪 高频震动切片机 活体循环系统 AMIRA三维视图和分析软件 高速固态硬盘
- 透明化试剂盒系列
- 组织透明化试剂盒(亲水型) 组织膨胀试剂盒 胚胎透明化试剂盒 增强型组织透明化试剂盒 细胞膨胀试剂盒 皮肤透明化试剂盒 组织切片透明&膨胀试剂盒(试用装) 骨组织透明化试剂盒 上皮或结缔类组织透明化试剂盒 非固定形态类软组织透明化试剂盒 肿瘤或致密组织透明化试剂盒 颅骨-脑组织透明化试剂盒 植物透明化试剂盒 活体颅骨透明化试剂盒 整鼠透明化试剂盒 Lectin血管标记物 PBS缓冲液 4%多聚甲醛固定液(PFA)
- 组织透明化及成像服务
- 单细胞空间多组学与高通量蛋白质组学科研检测服务 一站式科研服务——组织透明化、免疫染色、3D成像、数据分析及存储
- 小动物活体成像
- 小动物近红外二区成像
- 纳米药物制备系统/服务
- 微流控纳米药物递送平台 纳米药物载体合成服务
- 超快超分辨功能超声成像系统
- 超快超分辨功能超声成像系统
- 活体多光子及高分辨显微成像系统
- 高分辨率显微成像系统 双光子荧光寿命计数系统 活体多光子显微成像系统
- 试剂耗材
- 抗体系列产品 GATTA 显微镜纳米标尺 RISystem小鼠骨钉 Cellendes细胞水凝胶
- 小动物灌注/灌流设备
- 小动物灌注系统 灌流设备及附件
- 类器官3D细胞培养
- 锘海类器官、3D细胞培养系统
- 生物3D打印设备
- RegenHU生物3D打印机
-
应用案例
- 成像视频
- 成像视频
- 成像图片
- 成像图片
- 新闻资讯
- 联系我们
光片技术小课堂(番外)—— 平铺光片技术
发布日期:2021/3/10 11:39:02
之前说到光片显微镜以其较高的三维空间分辨率、良好的光学切片能力和较高的成像速度而备受关注。但是由于衍射极限导致的光片形状限制(见光片技术小课堂二2.1),当需要大视场时,会导致较低的轴向分辨率或较差的光学切片能力。因此,如何产生出厚度均匀且薄,又大到能覆盖视场的激发光片成为光片显微镜的关键问题。目前已经有很多不同的方法来尝试在光片显微镜中产生这样的光片,例如高斯光片、贝塞尔光片和晶格光片。然而无论采用何种方法,这种光片自身特性的相互限制对所有类型的光片都是存在的,而且当我们需要用亚微米厚度的光片去对几十微米或更大的视场进行成像时,这个问题尤其具有挑战性。

Dean和Fiolka首先提出了一种解决方案,通过使用聚焦可调的透镜沿光的传播方向扫描激发光的焦点,从而可以创建长而均匀的虚拟激发光束,并且可以通过扫描虚拟激发光束进一步生成大而均匀的虚拟激发光片。但是这种方法有两个局限性,首先是由于扫描过程中激发光束尾迹的混合使得用这种方法产生的光片对激发光的限制很差,因此光学切片的能力较差,需要与共焦检测一起使用以抑制荧光背景;其次,由于产生的激发光片是一个虚拟光片,因此在一个相机曝光周期内,焦点需要扫描整个视场,因此在视场内任何特定位置的实际曝光时间都非常短,需要使用相对较高的激发功率才能产生足够的荧光信号,从而导致更强的光毒效应。
在这之后,西湖大学的高亮博士提出了一种在不增加光片厚度或降低光约束力的情况下提高光片显微镜视场的替代方案。如图1所示,可以通过沿光片长度方向在检测光路的焦平面内平铺光片并在每个位置拍摄图像来代替使用较大的激发光片对大视场进行成像。然后,将所有图像组合在一起,重建整个视场的图像。尽管这种方法降低了成像速度,但其所带来的好处是显而易见的,采用更薄的光约束能力越好的光片对同一样品进行成像,可以获得更高的轴向分辨率和更好的光学切片能力。
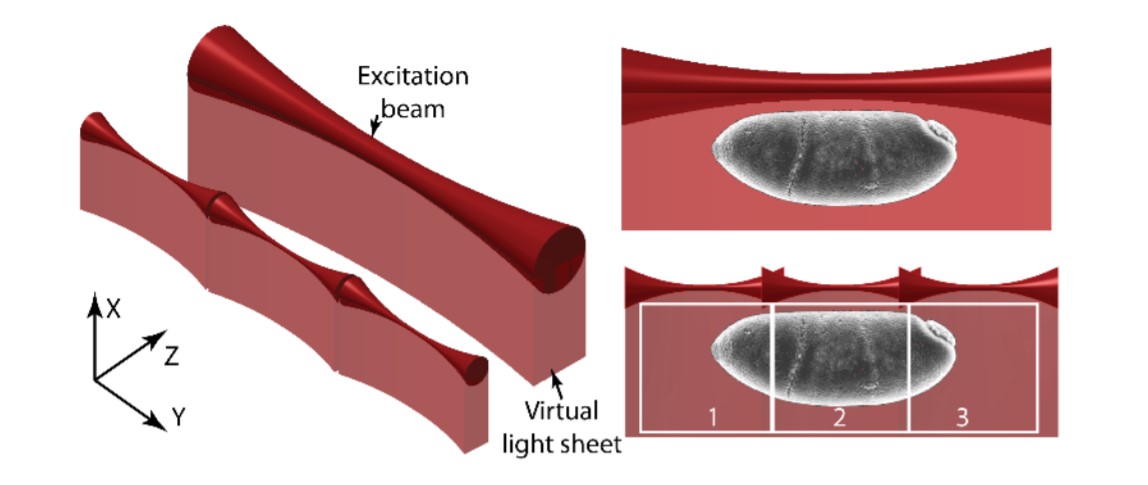
图1 该图展示了该方法的工作原理,同样的视野可以通过沿着光片长度方向平铺更小但更薄的光片并拍摄额外的图像来成像。
为了在实际中实现这一方法,在成像过程中样品或激发光束必须沿光束方向快速移动。高亮博士选择通过使用纯相位空间光调制器(spatial light modulate, SLM)对光束进行离焦来移动激发光束,因为无论是移动样品还是激发物镜都太慢。此外由于光片显微镜中激发物镜和检测物镜之间的物理约束,激发物镜的NA通常小于0.6,因此激发光束可以在对其横向光强分布影响很小的情况下离焦几百微米。
接下去高亮博士在前期严格的数字模拟后通过两个实际成像实验验证该方法的可行性,首先
是用1.5%琼脂糖凝胶包埋100nm黄绿色荧光粒子来验证该方法的成像性能,结果如下:图2(a) – 2(c)展示了在三个不同激发光束位置(左、中、右)下为同一视场采集的三个三维图像堆栈的轴向更大值投影;图2(d) – 2(f)展示了相应的去卷积图像结果;图2(h) – 2(m)展示了选定的相同荧光粒子在去卷积前后的高分辨率对比视图,以及每个选定粒子的轴向强度图。
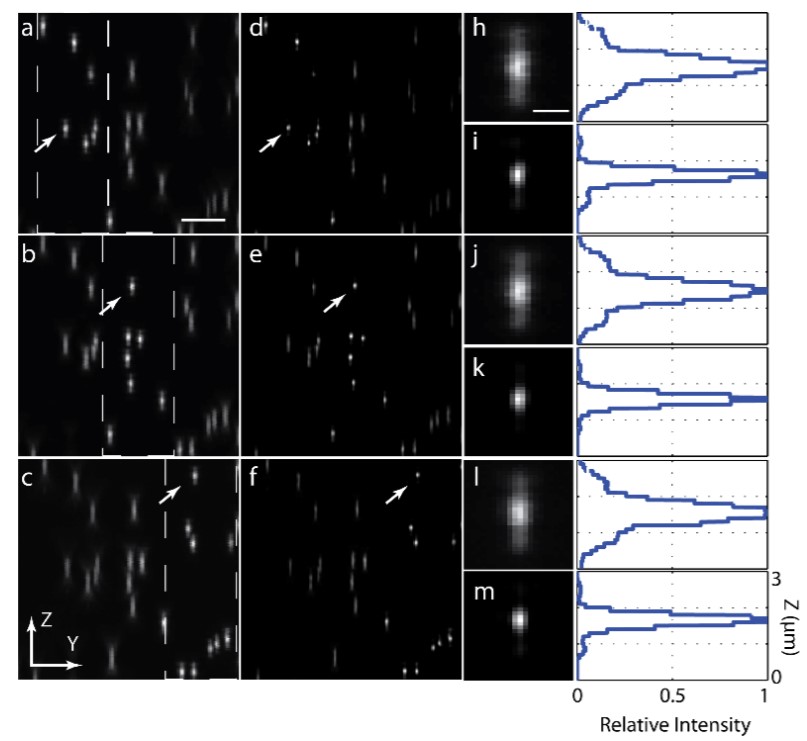
图2 用平铺光片方法对嵌入琼脂糖凝胶中的荧光粒子进行三维成像。(a-c) 在激发光片分别位于视场的左侧(a)、中间(b)和右侧(c)时的同一视场下的YZ更大值投影;(d-f) YZ去卷积后的更大值投影;(h-m) 同一视场不同区域中所选荧光粒子的放大视图与轴向强度图;比例尺:a为5μm,h为1μm。
如图2所示,在每个光束位置由于与视场相比激发光束长度更短,因此在相应的子区域中获得了比其余区域更高的轴向分辨率,通过在三个位置平铺激发光片,可以在整个视场内获得~500nm的均匀轴向分辨率。
第二个实验通过对在3折叠期的活体线虫胚胎成像进一步证明了其成像性能。在这个阶段,胚胎内大约有550个细胞,这种胚胎的细胞结构非常致密,它会引起强烈的光学像差和散射。为了获得具有亚微米轴向分辨率和高信噪比的三维图像,需要具有良好的光学切片能力。在三个位置之间移动相同的激发光束以成像整个胚胎,胚胎的横向更大值投影如图3(a)所示。为了更好地揭示胚胎的内部细胞结构,图3(b) – 7(d)展示了在不同光束位置下沿水平虚线的胚胎截面图,图3(e) – 3(g)展示了沿三条垂直虚线的胚胎截面图。尽管胚胎的结构复杂,光学像差大,但由于使用了具有良好光约束力的薄激发光束,通过移动激发光束并拍摄额外的图像,整个胚胎能够如预期那样以~500nm的轴向分辨率和高信噪比成像。
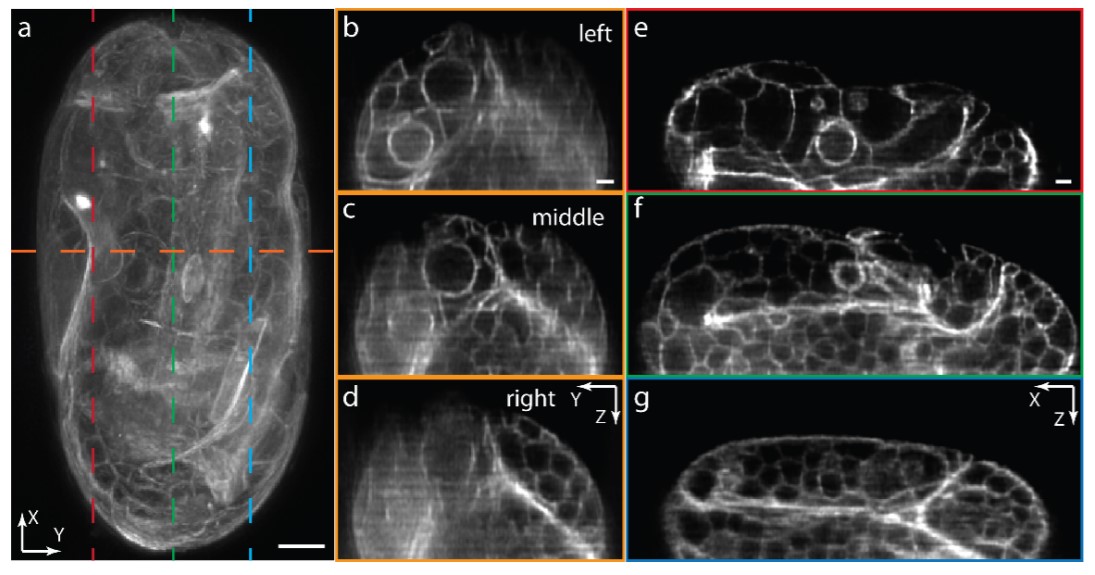
图3 用平铺光片方法对3折叠期线虫胚胎进行了三维成像。(a) 胚胎XY更大值投影;(b-d) 在激发光片位于胚胎的左侧(b)、中间(c)和右侧(d)时胚胎沿(a)所示水平线的YZ截面图;(e-g) 沿(a)所示三条垂直线处的胚胎XZ截面图。比例尺:a为5μm,b和e为2μm。
高亮博士在该研究中提出并展示了一种新型光片显微镜进行的三维成像方法,能够在获得高三维空间分辨率和良好的光学切片能力的同时获得大视场。与以前实现这一目标的解决方案相比,它没有试图扩展光片的尺寸至足够覆盖整个视场,而是通过使激发光束离焦并拍摄同一图像平面的多个图像,将相对较小的激发光片平铺到多个位置。通过这个方法,在只降低成像速度的情况下能获得具有更薄厚度和更好的光约束力的光片。高亮博士提出的方法是很有价值的,首先,尽管已经引入了不同种类的光片来提供对该问题的解决方案,但这些方案无论是使用什么方法产生的激发光片,仍普遍是将光片变得更厚或者增加光片的尺寸导致光约束力(即光学层切能力)变得更差,这样的思路由于光的衍射极限的限制,不可能有更好的解决方案。因此,该方法是在轴向分辨率、光学切片能力和视场均不受影响的前提下,提供的一种新的策略。其次,所提出的方法对任何类型的光片显微镜都是有效的,这意味着使用同一种尺寸较小、厚度较薄且光约束能力较好的光片,可以从同一样品中获得更高的轴向分辨率和更好的光学切片能力。
锘海LS18平铺光片显微镜正是使用了这一精妙的技术作为产品的核心技术,旨在为广大生命科学研究者提供更好更便捷的成像工具及成像技术。

锘海LS18平铺光片显微镜
专为透明化样品设计的高速高分辨三维荧光显微成像系统
参考文献:(点击“阅读原文”参看文献)
1. Liang.Gao, “Extend the field of view of selective plan illumination microscopy by tiling the excitation light sheet,” Optics Express 23(5), 6102-11 (2015).
2. J. Huisken, J. Swoger, F. Del Bene, J. Wittbrodt, and E. H. K. Stelzer, “Optical sectioning deep inside live embryos by selective plane illumination microscopy,” Science 305(5686), 1007–1009 (2004).
3. P. J. Keller, A. D. Schmidt, J. Wittbrodt, and E. H. K. Stelzer, “Reconstruction of zebrafish early embryonic development by scanned light sheet microscopy,” Science 322(5904), 1065–1069 (2008).
4. T. A. Planchon, L. Gao, D. E. Milkie, M. W. Davidson, J. A. Galbraith, C. G. Galbraith, and E. Betzig, “Rapid three-dimensional isotropic imaging of living cells using Bessel beam plane illumination,” Nat. Methods 8(5), 417–423 (2011).
5. L. Gao, L. Shao, C. D. Higgins, J. S. Poulton, M. Peifer, M. W. Davidson, X. Wu, B. Goldstein, and E. Betzig, “Noninvasive imaging beyond the diffraction limit of 3D dynamics in thickly fluorescent specimens,” Cell 151(6), 1370–1385 (2012).
6. Y. Wu, A. Ghitani, R. Christensen, A. Santella, Z. Du, G. Rondeau, Z. Bao, D. Colón-Ramos, and H. Shroff, “Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans,” Proc. Natl. Acad. Sci. U.S.A. 108(43), 17708–17713 (2011).
7. B.-C. Chen, W. R. Legant, K. Wang, L. Shao, D. E. Milkie, M. W. Davidson, C. Janetopoulos, X. S. Wu, J. A. Hammer 3rd, Z. Liu, B. P. English, Y. Mimori-Kiyosue, D. P. Romero, A. T. Ritter, J. Lippincott-Schwartz, L. Fritz-Laylin, R. D. Mullins, D. M. Mitchell, J. N. Bembenek, A. C. Reymann, R. B?hme, S. W. Grill, J. T. Wang, G. Seydoux, U. S. Tulu, D. P. Kiehart, and E. Betzig, “Lattice light-sheet microscopy: Imaging molecules to embryos at high spatiotemporal resolution,” Science 346(6208), 1257998 (2014).
8. U. Krzic, S. Gunther, T. E. Saunders, S. J. Streichan, and L. Hufnagel, “Multiview light-sheet microscope for rapid in toto imaging,” Nat. Methods 9(7), 730–733 (2012).
9. R. Tomer, K. Khairy, F. Amat, and P. J. Keller, “Quantitative high-speed imaging of entire developing embryos with simultaneous multiview light-sheet microscopy,” Nat. Methods 9(7), 755–763 (2012).
10. J. Durnin, “Exact solutions for nondiffracting beams. I. The scalar theory,” J. Opt. Soc. Am. 4(4), 651–654 (1987).
11. K. M. Dean and R. Fiolka, “Uniform and scalable light-sheets generated by extended focusing,” Opt. Express 22(21), 26141–26152 (2014).
12. R. Fiolka, A. Stemmer, and Y. Belyaev, “Virtual slit scanning microscopy,” Histochem. Cell Biol. 128(6), 499–505 (2007).
13. F. O. Fahrbach and A. Rohrbach, “Propagation stability of self-reconstructing Bessel beams enables contrastenhanced imaging in thick media,” Nat. Commun. 3, 632 (2012).
14. L. Shao, P. Kner, E. H. Rego, and M. G. L. Gustafsson, “Super-resolution 3D microscopy of live whole cells using structured illumination,” Nat. Methods 8(12), 1044–1046 (2011).
